728x90
반응형
miRNA 발현 데이터를 분석하는 R 코드를 정리해보았습니다.
0. 필요 함수 불러오기
library(readxl)
library(clusterProfiler)
library(tidyverse)
1. 데이터 불러오기
setwd("C:/Users/abc/Desktop") # 작업 디렉토리 설정
설명:
- setwd() 함수는 작업 디렉토리를 설정하는 함수입니다.
- 데이터를 불러올 디렉토리를 지정해야 파일을 찾을 수 있습니다.
- 일반적으로는 같은 위치에 저장된 파일은 그냥 불러올 수 있습니다.
data <- read.csv("miRNA_data.csv", header=TRUE) # CSV 파일 불러오기
설명:
- read.csv() 함수는 CSV 파일을 읽어 데이터프레임으로 변환합니다.
- header=TRUE 옵션은 첫 번째 행을 열 이름으로 사용하도록 지정합니다.
2. 그룹 나누기
Group1 <- data[,2:4] # 첫 번째 그룹 (예: Control 그룹)
Group2 <- data[,5:7] # 두 번째 그룹 (예: Treatment 그룹)
설명:
- 데이터프레임의 특정 열을 선택하여 두 개의 그룹을 만듭니다.
- data[,2:4]은 2~4번째 열을 선택하여 Group1에 저장합니다.
- data[,5:7]은 5~7번째 열을 선택하여 Group2에 저장합니다.
3. 평균 및 Fold Change 계산
mean_Group1 <- apply(Group1, 1, mean) # Group1의 각 행 평균 계산
mean_Group2 <- apply(Group2, 1, mean) # Group2의 각 행 평균 계산
설명:
- apply(데이터, MARGIN, 함수)는 행 또는 열에 함수를 적용하는 함수입니다.
- MARGIN=1은 행별 연산을 의미합니다. (열별 연산은 MARGIN=2)
- mean을 사용하여 각 miRNA의 평균 발현 값을 계산합니다.
- rowMeans(df[,2:4])를 사용해도 됩니다.
log2FC <- log2(mean_Group2 / mean_Group1) # 로그2 변환된 Fold Change 계산
설명:
- Fold Change는 그룹 간 평균 발현량의 비율을 의미합니다.
- log2()를 사용하여 로그2 변환된 Fold Change 값을 계산합니다.
4. 통계 분석 (T-test)
p_values <- apply(data[,2:7], 1, function(x) t.test(x[1:3], x[4:6])$p.value)
설명:
- apply()를 사용하여 각 miRNA에 대해 t-검정을 수행합니다.
- t.test(x[1:3], x[4:6])는 두 그룹 간의 평균 차이를 검정하는 함수입니다.
- $p.value를 사용하여 p-value만 저장합니다.
- var.equal=FALSE가 일반적으로 디폴트 값인데, =TRUE로 놓으면 등분산가정 t검정을 수행한 결과를 보여주게 됩니다. *예시: t.test((df[,2:4][i,]),(df[,5:7][i,]), var.equal=TRUE)$p.value
adjusted_p <- p.adjust(p_values, method="BH") # 다중 검정 보정
설명:
- p.adjust() 함수는 다중 검정을 보정하는 함수입니다.
- "BH" (Benjamini-Hochberg) 방법을 사용하여 FDR(거짓 발견율)을 조정합니다.
- 데이터 및 분석 방향에 따라서 생략해도 되는 부분인 것 같습니다.
반응형
5. Volcano Plot 시각화
log_p <- -log10(adjusted_p) # -log10 변환된 p-value 계산
설명:
- 작은 p-value를 강조하기 위해 -log10() 변환을 수행합니다.
library(ggplot2) # ggplot2 패키지 로드
설명:
- ggplot2는 R에서 가장 널리 사용되는 데이터 시각화 패키지입니다.
data$color <- ifelse(adjusted_p < 0.05 & abs(log2FC) > 1, "red", "black")
설명:
- ifelse()를 사용하여 p-value가 0.05 미만이고 log2FC가 1 이상이면 빨간색, 그렇지 않으면 검은색을 지정합니다.
ggplot(data, aes(x=log2FC, y=log_p, color=color)) +
geom_point() +
theme_minimal() +
labs(title="Volcano Plot", x="Log2 Fold Change", y="-Log10 p-value")
설명:
- aes(x=log2FC, y=log_p, color=color)를 통해 시각화할 축과 색상을 설정합니다.
- geom_point()는 점 그래프를 생성합니다.
- theme_minimal()을 사용하여 깔끔한 스타일의 그래프를 만듭니다.
- labs()를 사용하여 그래프의 제목과 축 이름을 설정합니다.
실제로 제가 수행할때에는 중간중간 찾으며 추가를 해서 아래와 같이 작성했던 것 같습니다.
df_Fn[(df_Fn$Fold_change_Fn >= 2) & (df_Fn$p_value_Fn <= 0.05), "Fn"] = "Upregulated"
df_Fn[(df_Fn$Fold_change_Fn <= 0.5) & (df_Fn$p_value_Fn <= 0.05), "Fn"] = "Downregulated"
ggplot(data=df_Fn, aes(x=log_Fold_change_Fn, y=log_p_value_Fn)) +
xlab(expression(Log[2] ~ (Fold ~ Change))) +
ylab(expression(-Log[10] ~ (P-value))) +
scale_colour_manual(values = c("blue", "darkgray", "red")) +
geom_vline(xintercept = 1, linetype = 'dashed', col = 'gray', size = 1) +
geom_vline(xintercept = -1, linetype = 'dashed', col = 'gray', size = 1) +
geom_hline(yintercept = 1.30103, linetype = 'dashed', col = 'darkgray', size = 1) +
geom_point(aes(colour = Fn)) +
theme(text = element_text(size = 17))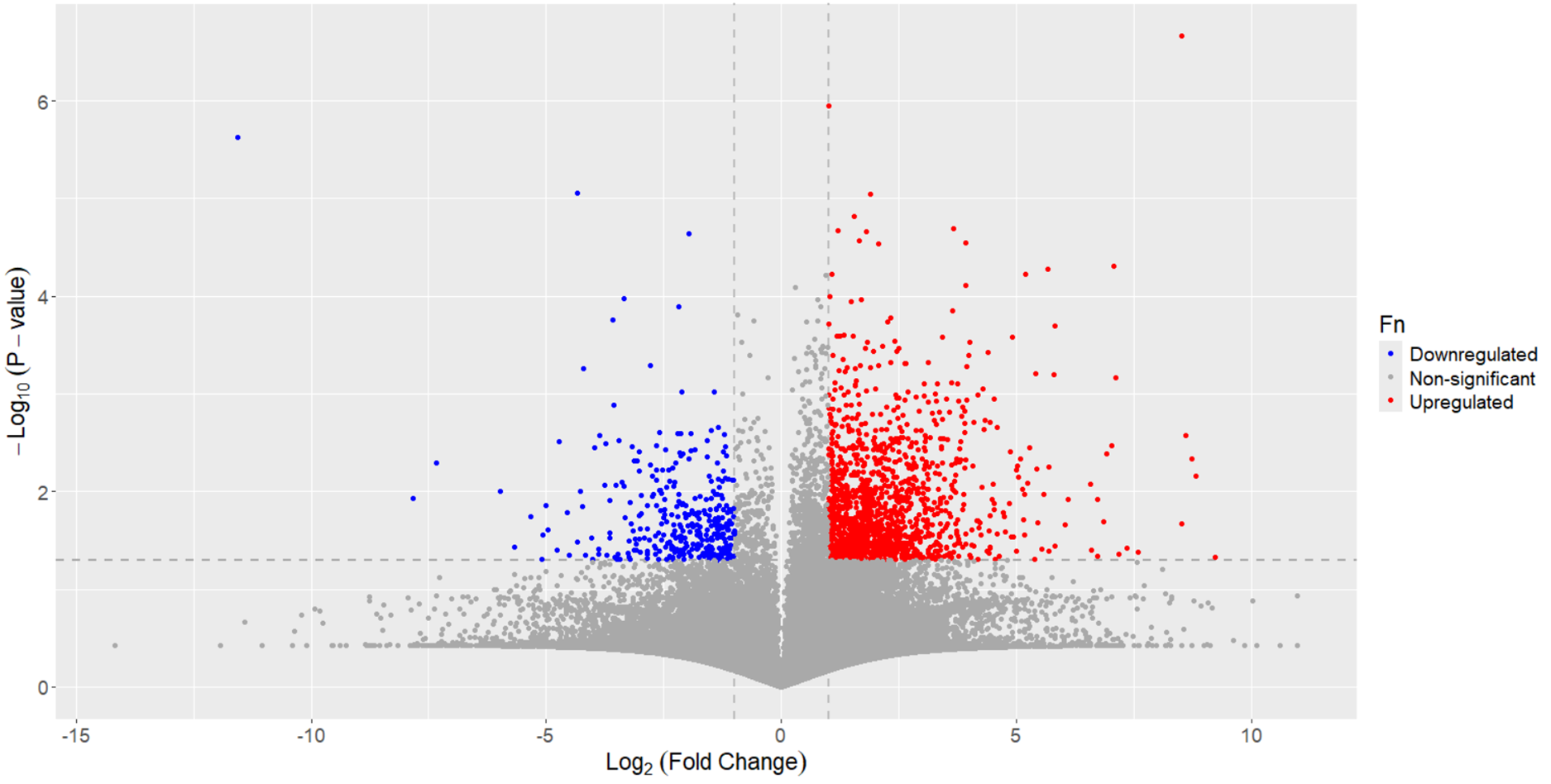
6. 결과 저장
write.csv(data, "miRNA_analysis_results.csv", row.names=FALSE)
설명:
- write.csv()를 사용하여 분석 결과를 CSV 파일로 저장합니다.
- row.names=FALSE 옵션은 행 이름을 저장하지 않도록 설정합니다.
엑셀 형태로 저장 시에는 아래와 같이 하게 됩니다.
library(writexl)
write_xlsx(df_Fn, "result.xlsx")
728x90
반응형
'R note' 카테고리의 다른 글
| R 데이터 프레임에서 특정 열 값 변환하기 (0) | 2025.03.13 |
|---|---|
| R에서 t.test() 오류 해결: "데이터는 본질적으로 상수입니다" (0) | 2025.03.10 |
| Heatmap, GO Plot, KEGG Plot (0) | 2025.02.01 |
