[DAVID: Functional Annotation Bioinformatics Microarray Analysis]
DAVID (Database for Annotation, Visualization and Integrated Discovery)는 연구자들이 대규모 유전자 목록의 생물학적 의미를 이해할 수 있도록 돕는 포괄적인 기능 주석 도구 세트를 제공하고 있습니다. 이 도구들은 여러 출처의 기능 주석을 통합하는 DAVID Gene 개념을 기반으로 구축된 DAVID Knowledgebase에 의해 지원됩니다.
특정 유전자 목록에 대해 DAVID 도구는 아래와 같은 수행을 할 수 있습니다.
- 풍부한 생물학적 테마 확인: 특히 Gene Ontology (GO) 용어
- 기능 관련 유전자 그룹 발견
- 중복된 주석 용어 클러스터링
- BioCarta 및 KEGG 경로 맵에서 유전자 시각화
- 다수 유전자-다수 용어 관련 2차원 보기 표시
- 목록에 없는 기능적으로 관련된 다른 유전자 검색
- 상호작용하는 단백질 목록화
- 유전자 이름 일괄 탐색
- 유전자-질병 연관 링크
- 단백질 기능 도메인 및 모티프 강조 표시
- 관련 문헌으로 리디렉션
- 유전자 식별자를 다른 유형으로 변환
- 그 외 다양한 기능
이 도구들은 연구자들이 대규모 유전자 목록의 생물학적 맥락을 심도 있게 탐구할 수 있도록 다양한 분석 기능을 제공하고 있습니다.
DAVID의 주소는 아래와 같습니다.
https://david.ncifcrf.gov/
DAVID Functional Annotation Bioinformatics Microarray Analysis
david.ncifcrf.gov
들어가면 아래와 같은 페이지를 볼 수 있습니다.

여기에서, gene 관계 분석을 수행하기 위해 'Shortcut to DAVID Tools'에서 'Functional Annotation'으로 들어가봅니다.

그럼, 아래와 같은 페이지를 볼 수 있습니다.

왼쪽의 Step1에는 원하는 Protein Coding gene의 이름을 적어줍니다. (여러개 가능합니다.)
Step2에서는 'OFFICIAL_GENE-SYMBOL'을 택해준 후, 아래에 'Homo sapiens'를 찾아줍니다. (사람의 유전자를 적었기 때문에 그렇습니다.)

그럼, 다음과 같이 입력한 gene들에 관한 분석 결과를 보여줍니다.

예를들어, Clustering 부분을 클릭하면 아래와 같이 나오는데, 유사한 생물학적 의미를 가진 gene기리 묶어서 표시해준 것으로 보입니다.
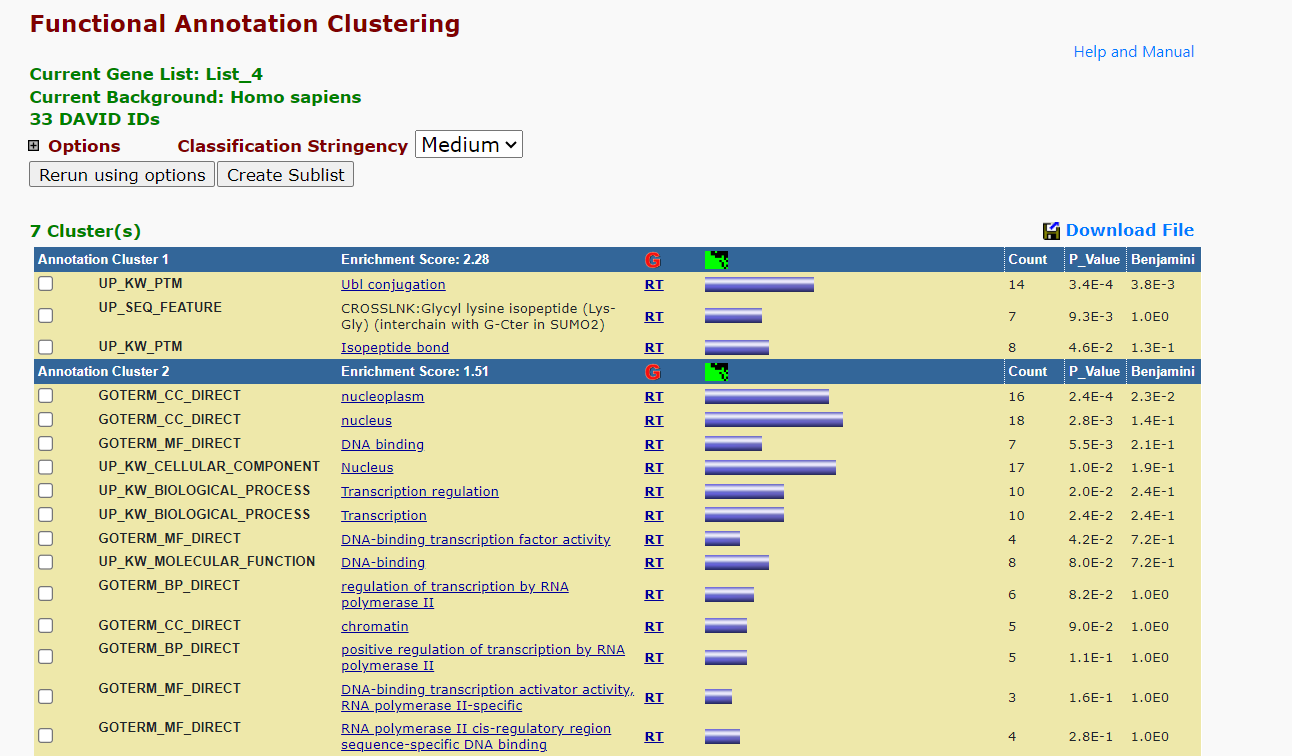
여기에서 초록색 그림을 누르면, 아래처럼 2D View를 보여줍니다.
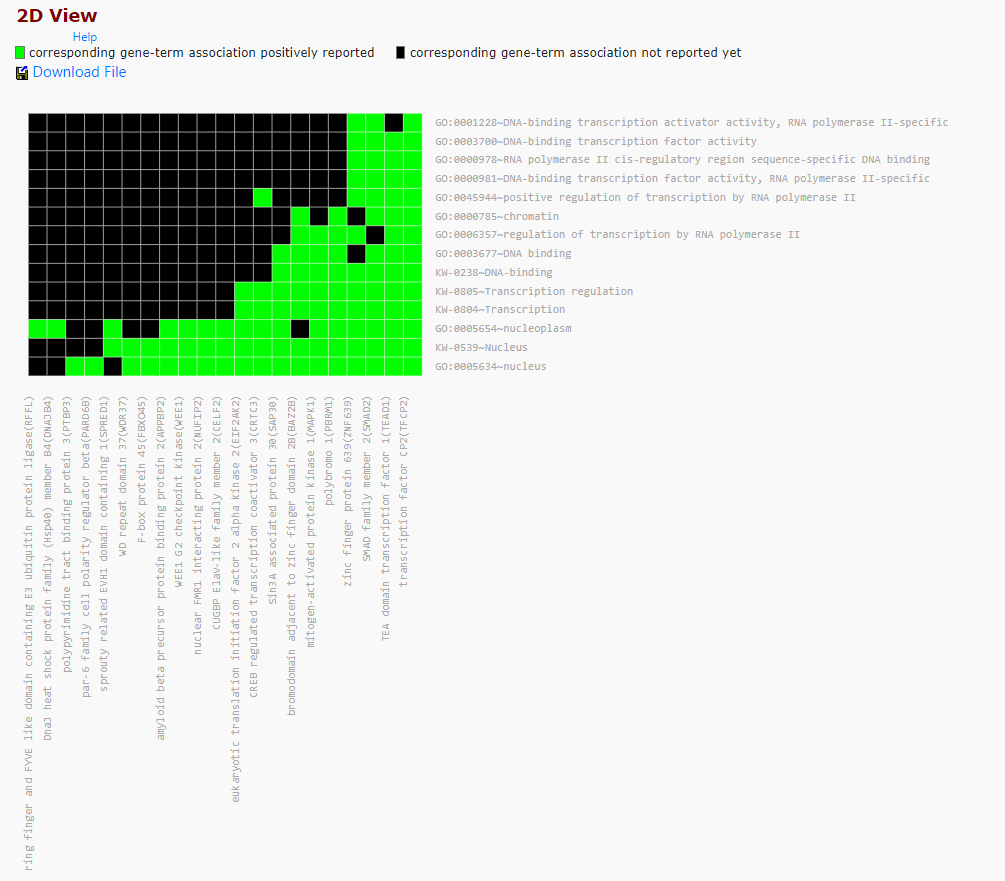
이 외에도, 여러가지 기능과 정보를 제공하는 것으로 보입니다.
저도 추가적인 공부가 필요할 것 같습니다.
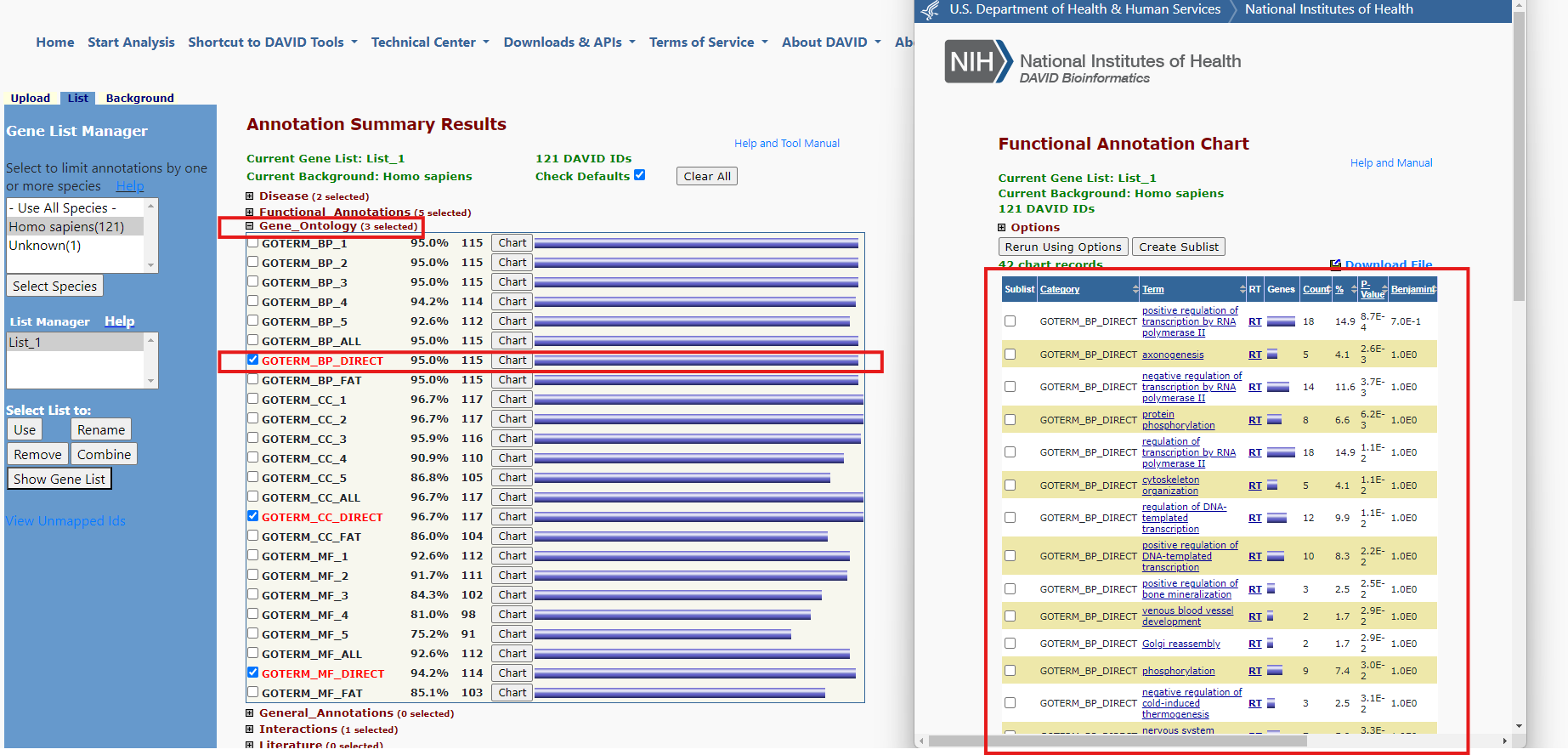
<GO 분석 결과>
아래처럼, 분석 결과 목록 중 gene ontology를 누르면 BP/CC/MF에 대한 결과를 볼 수 있습니다. 원하는 항목을 누르면, 오른쪽과 같이 GO분석 결과를 보여줍니다.
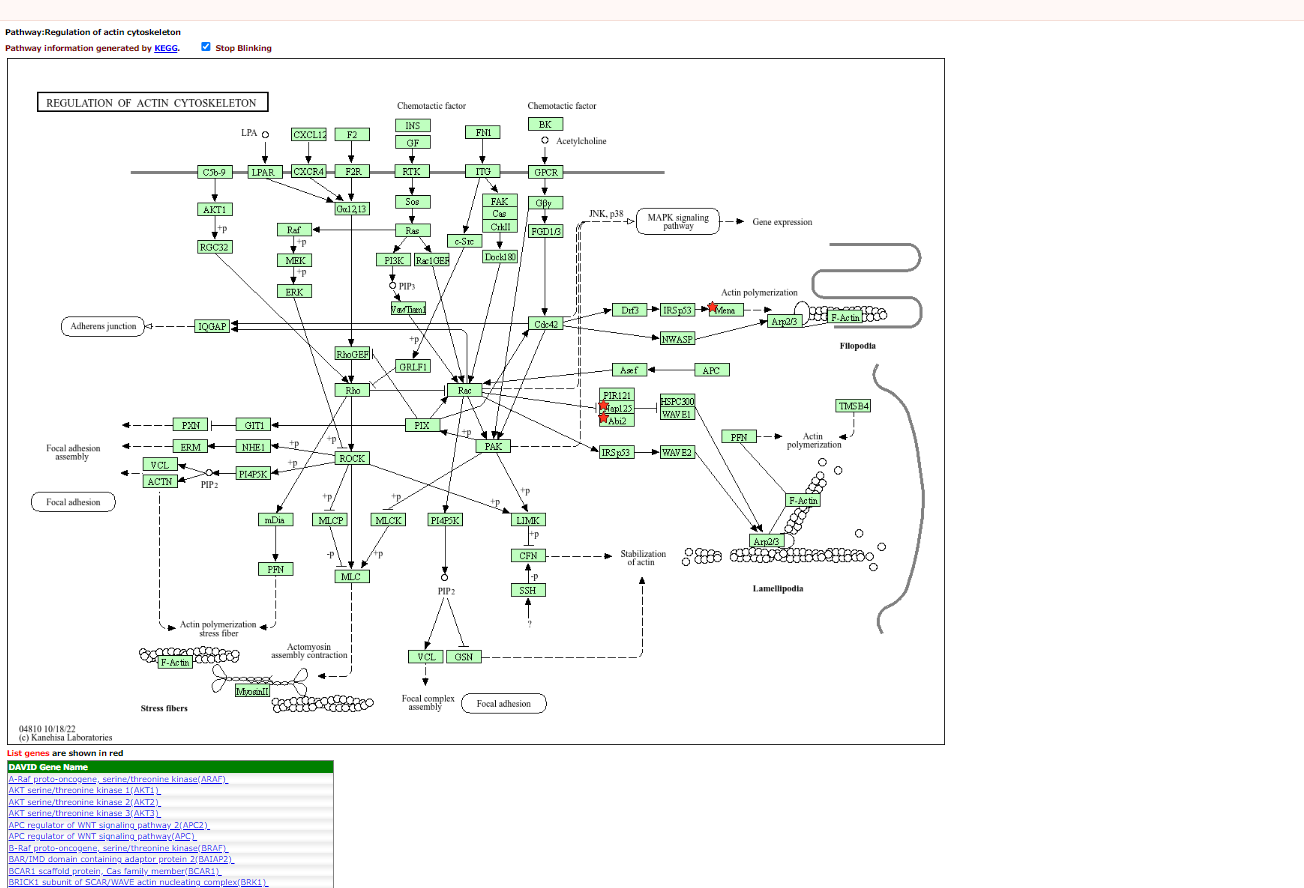
<KEGG 분석 결과>
아래처럼, KEGG pathway를 누르면 분석된 pathway를 볼 수 있습니다.
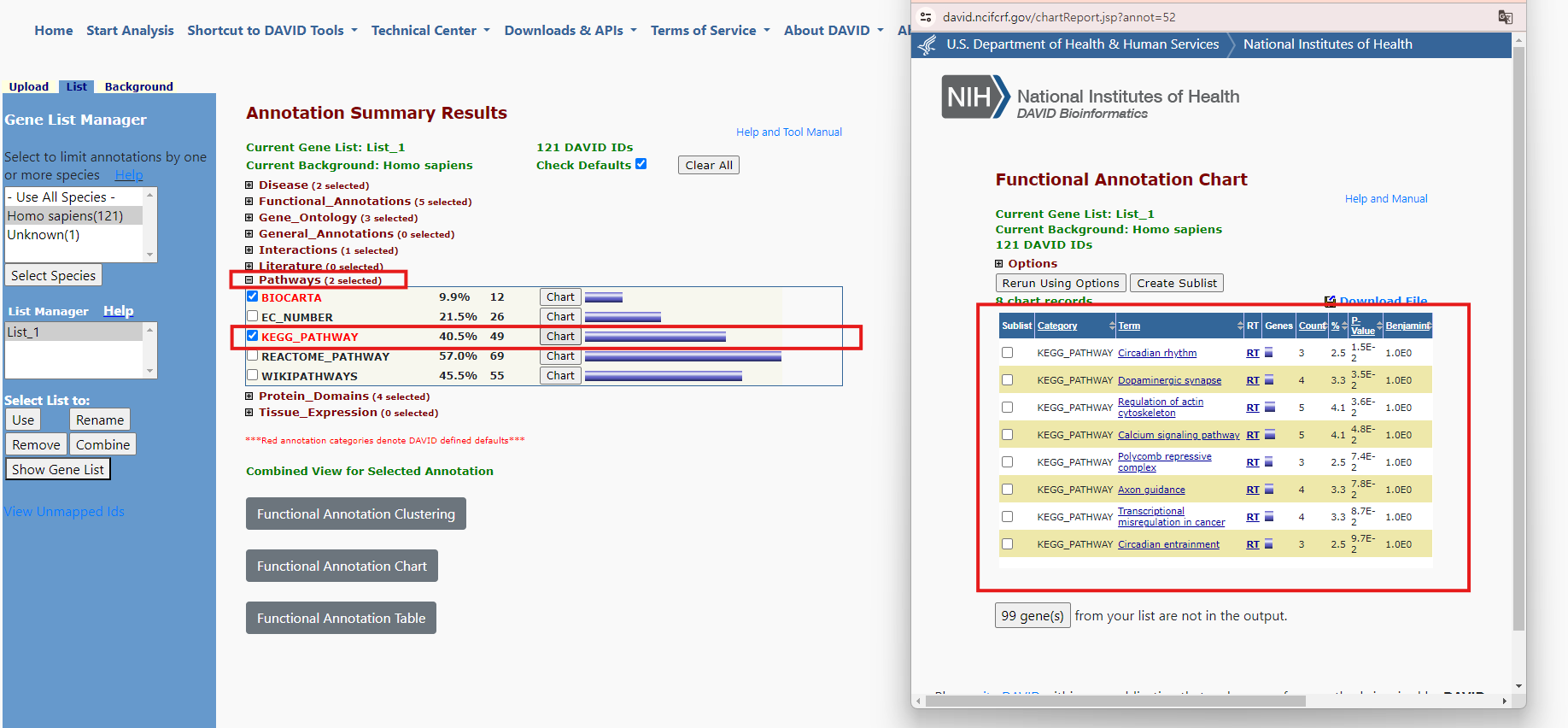
Chart를 보면 아래와 같이 자세한 정보를 보여줍니다.
'생물정보학' 카테고리의 다른 글
| bioDBnet - Gene ID 변경하기 (type 변경) (0) | 2024.07.19 |
|---|---|
| circRNA의 Target gene 찾기 (CircInteractome) (2) | 2024.07.18 |
| Primer 짜는 방법 (NCBI) (0) | 2024.07.15 |
| STRING - 단백질 상호작용 정보 데이터베이스 (0) | 2024.07.08 |
| TargetScan - miRNA Target gene 찾기 (생물학적 표적 예측 서버) (4) | 2024.07.05 |



